久しぶりに VPSの環境を変更したら結構苦労した
私はConoha VPSというサービスを使って、2017年頃からブログを運営しています。これまでは非常に満足していたのですが、最近になってVPS自体がアップグレードされ、新しいバージョン3がリリースされていることに気づきました。そこで、この新しいバージョンを使って、費用を少し抑えつつ環境を移行しようと考えました。
私が運営しているのは3つのサイトで、これらをすべて新しい環境に移行しようとしたのですが、思っていた以上に苦労しました。その中でも特に大変だったのが、VPSに追加されたセキュリティグループの設定です。この設定のおかげで、何と2時間も手間取ってしまいました。
具体的には、セキュリティグループの設定でHTTPやHTTPS、SSHのポートが開放されていないと、サイト構築がうまくいかないことが分かりました。私の方でもターミナルを使って内部からポートが開いていることを確認していたのですが、問題はそれだけではありませんでした。どうやら、VPSのさらに外側にセキュリティポリシーが存在し、それを適切に設定する必要があったようです。
これぐらい開けとかないとまともにサイト運営できないですね。AWSとかだとこういうの普通ですけど、以前のVPSではこんなのなかったのでちょっと面食らいました。
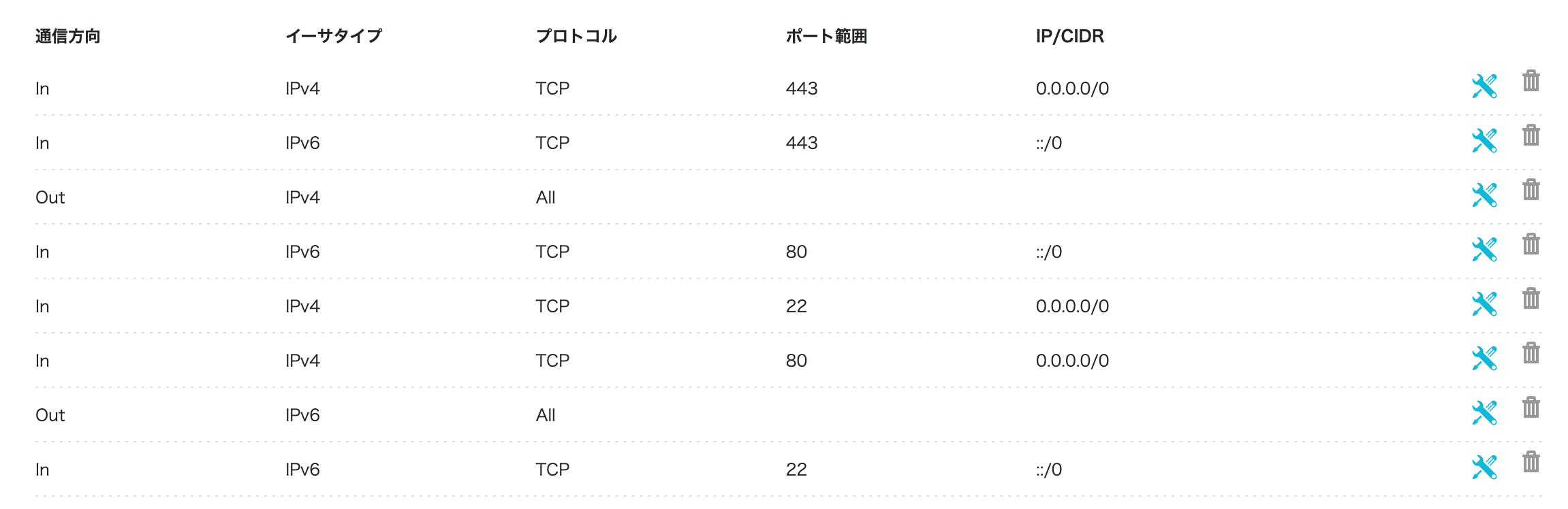
今回の経験から、移行時にはVPSの外部・内部両方のセキュリティ設定をきちんと確認することの重要性を改めて実感しました。これからConoha VPSを使って新しいバージョンに移行する方がいれば、同じようなトラブルを避けるために、セキュリティグループの設定に十分注意することをお勧めします。
firewall-cmd
便利コマンドですね。
firewall-cmd --list-all ・・・・・・ services: cockpit dhcpv6-client http https ssh ports: 443/udp
こんな感じだと問題なくインスタンス自体のポートは開いています。
ports: 443/udp ですが、services の方にhttp, https, ssh が開いていますので80/tcpとか 443/tcpとか22/tcpは開いています。これでうまくいかないとすると、要するにセキュリティグループの設定側で閉じているということです。
KUSANAGIも変化していて苦労した
KUSANAGIさん変わりすぎ
kusanagiさんが変わりすぎているので、なかなか大変でした。
Basic認証って何やねん
ワードプレスが狙われすぎてセキュリティーが大変と言うことでこういう認証が取り入れられたんですね。
普通に使うのには面倒すぎる。
外したい時は、
/etc/opt/kusanagi/nginx/conf.d/applingo.wp.inc
の
auth_basic "basic authentication";
auth_basic_user_file "/home/kusanagi/.htpasswd";
削除するなり、コメントアウトする必要があります。
KUSANAGIさんアップロードの最大値低すぎ



コメント